[Deep Learning 5] Adam과 Xavier/He 초깃값 등(MNIST 활용)
SGD를 개선한 Optimizer
먼저 SGD(확률적 경사하강법)의 간략적인 코드부터 살펴보자
# SGD 클래스 구현
# 이번 장은 SGD의 단점을 개선한 클래스들을 구현해간다
class SGD:
def __init__(self, lr=0.01):
self.lr = lr
def update(self, params, grads):
for key in params.keys():
params[key] -= self.lr * grads[key]
학습률과 그래디언트를 곱해 매개변수들을 갱신해가는, 계속 다루고 있는 형태다.
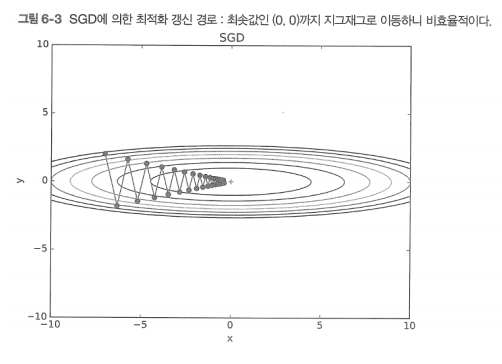
minimum을 찾아가는 경로를 보면 GD(경사하강법)가 더 나아 보이지만, GD는 계산이 느리다. 하지만 SGD도 각 등고선의 수직 방향으로 나아가기 때문에, 지그재그 모양으로 minimum을 찾아가며 시간이 많이 들고 비효율적인 경로를 보인다.
이는 특히 비등방성(anisotropy; 이방성) 그래프에서 쉽게 나타나는데, 축에 따라 기울기가 크게 다를 경우 SGD는 여러 곳을 왔다갔다하며 최적의 경로를 보이지 못한다. 그래서 나온 방식 중 하나가 모멘텀이다.
Momentum Optimizer
# 모멘텀 클래스 구현
# 종전의 속도(velocity, 방향성 있는 벡터)에 관성을 부여하며 갱신
import numpy as np
class Momentum:
def __init__(self, lr=0.01, momentum=0.9):
self.lr = lr
self.momentum = momentum # 모멘텀 변수: 종전의 속도를 거의 유지시킨다
self.v = None
def update(self, params, grads):
if self.v is None: # 아직 한 번도 갱신이 되지 않아 v(속도)가 없다면
self.v = {}
for key, val in params.items():
self.v[key] = np.zeros_like(val) # 형상에 맞게 0으로 초기화해 생성
for key in params.keys():
# 종전의 v의 90%에다가 -학습률*그래디언트를 합하여 벡터(행렬)의 합을 계산
# 새롭게 갱신된 v를 파라미터에 더함으로써 갱신
self.v[key] = self.momentum * self.v[key] - self.lr * grads[key]
params[key] += self.v[key]
모멘텀(Momentum) 방식은 물리에서의 ‘운동량’에서 따온 이름인데, 쉽게 말해 기존에 가던 경로에 관성을 주겠다는 의미이다.
\[v\leftarrow \alpha v-\eta \frac{\partial L}{\partial W}\] \[W\leftarrow W+v\]에타(eta) 외에도 알파(alpha) 값이 보이는데, 이건 모멘텀 변수(마찰 계수)이다. 종전에 움직이던 기울기에 알파(여기선 90%)를 곱해 거의 유지해주고, 거기다가 학습률*그래디언트를 더해주는 방식이다.
즉 최신 학습 과정에서 그래디언트가 갱신되었다고해서, 바로 그 방향으로 가는 것이 아니라, 종전에 가던 방향의 90%와 새로 계산된 그래디언트의 합벡터(행렬의 합) 방향으로 이동시키겠다는 의미이다. 그래서 관성, 속도라고 표현되는 듯
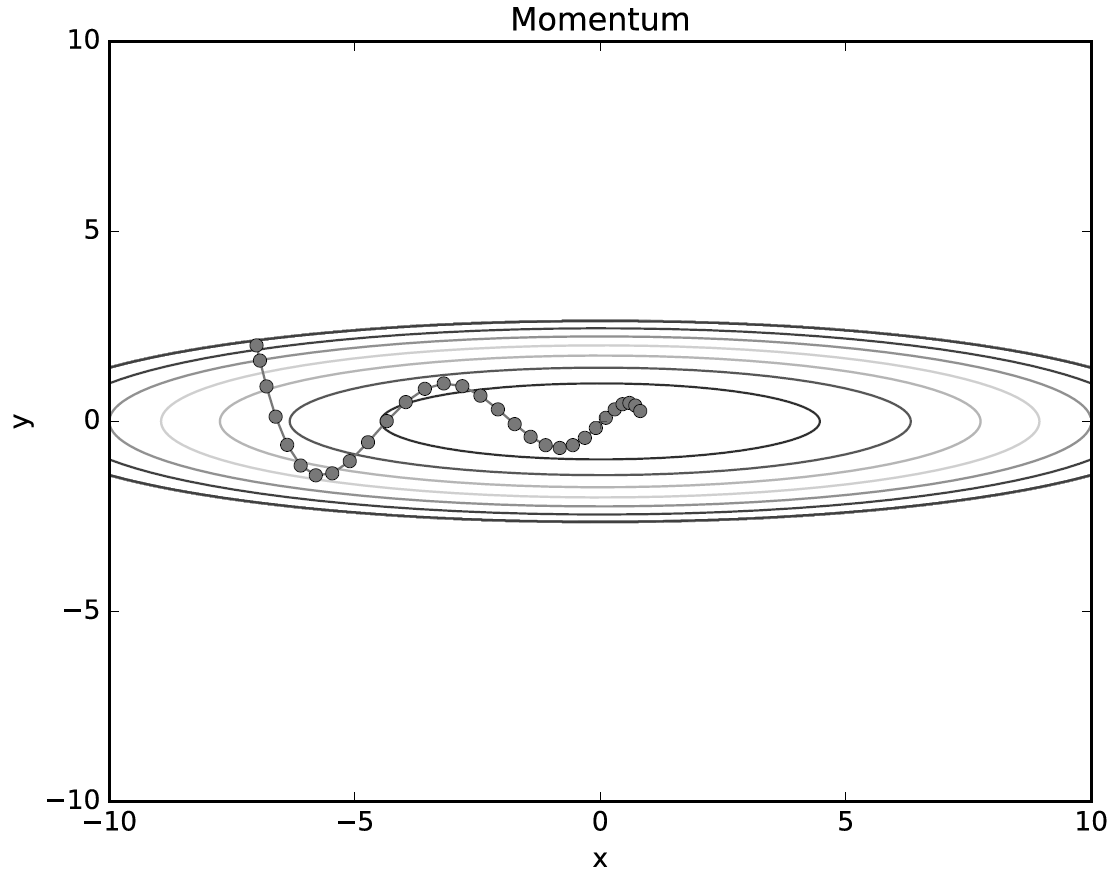
SGD처럼 딱딱하게 움직이는 것보다는, 비교적 출렁거리면서 관성을 유지하며 움직이는 것을 볼 수 있다. 그러나 이것도 양옆으로 와리가리가 많은 편. 더 좋은 모델은 없을까?
Momentum의 장단점 (*)
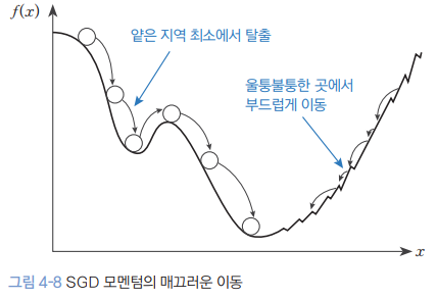
모멘텀은 관성이 있어, 특히 경사에서 내려올 경우 운동관성이 더 붙는다. 따라서 얕은 지역 최소(local minimum)에 빠지더라도 기존 GD(경사하강법)와 달리 무사히 빠져나가 전역 최소(global minimum)에 도달할 수 있다.
하지만 오버 슈팅(overshooting)이라는 단점도 있는데, 만약 전역 최소에 들어왔다고 하더라도 아직 운동관성이 붙어있어서 global minimum을 한 번 지나치게 된다는 문제가 있다.
이렇게 되면 최소점을 찾아도 속도가 떨어질 때까지 기다려야하는 단점이 발생한다.
AdaGrad(Adaptive Gradient) Optimizer
손실 함수의 경사가 가파를 때는 작은 폭으로 이동하고, 경사가 완만해지면 서서히 보폭(step size)을 늘리면서 유동적으로 minimum을 찾아가는 방식도 있다. 특히 더 가파른 곳에 위치한(=그래디언트가 큰) 원소일수록 다음 스텝은 더욱 조금만 움직이도록 제어하는 방식이다.
이를 AdaGrad(적응적 기울기; Adaptive Gradient) 방식이라고 하는데, 학습률 감소(learning rate decay)가 주 내용이다.
# AdaGrad(Adaptive Gradient) 클래스 구현
# 손실함수의 아다마르 곱을 h에 더한 후, h의 -1/2제곱을 학습률에 곱해준다
# 종전에 크게 움직인 원소는 이동 폭을 더 줄이겠다는 뜻인데, 무한히 학습하면 기울기가 0이 된다
class AdaGrad:
def __init__(self, lr=0.01):
self.lr = lr
self.h = None
def update(self, params, grads):
if self.h is None: # 아직 한 번도 갱신이 되지 않아 h가 없다면
self.h = {}
for key, val in params.items():
self.h[key] = np.zeros_like(val) # 형상에 맞게 0으로 초기화해 생성
for key in params.keys():
# 종전의 h에다가 학습률끼리 아다마르 곱을 하여 더한다
# 새롭게 갱신된 h에 루트를 씌워 학습률을 나눈다
self.h[key] += grads[key] * grads[key]
params[key] -= self.lr * grads[key] / (np.sqrt(self.h[key]) + 1e-7) # 1e-7은 dividedByZero 방지
AdaGrad의 가장 큰 특징은, 매개변수 전체의 학습률을 일괄적으로 낮추는 것이 아니라, ‘각각의’ 매개변수에 맞게 학습률을 개별적으로 낮춘다는 것에 있다. 코드로 이해가 안된다면 수식을 보면 된다.
\[h\leftarrow h+\frac{\partial L}{\partial W}\bigodot \frac{\partial L}{\partial W}\] \[W\leftarrow W-\eta \frac{1}{\sqrt{h}+\varepsilon } \frac{\partial L}{\partial W}\]먼저 변수 h는 각 단계에서 계산된 그래디언트의 아다마르 곱(Hadamard Product; 행렬의 각 원소별 곱셈)을 수행하는데, 이 과정에서 절댓값이 큰 그래디언트는 그렇지 않은 값들에 비해 제곱이 되며 더 커지게 된다.
그리고 이 커진 값에 루트를 씌워 그래디언트를 나눠주면, 비교적 컸던 그래디언트 원소는 더 큰 폭으로 줄어들게 되는 것이다. 즉 직전의 그래디언트가 클수록, 이번의 보폭은 더 큰 폭으로 줄여서 샅샅이 살펴보겠다는 의지가 반영된 식이다.
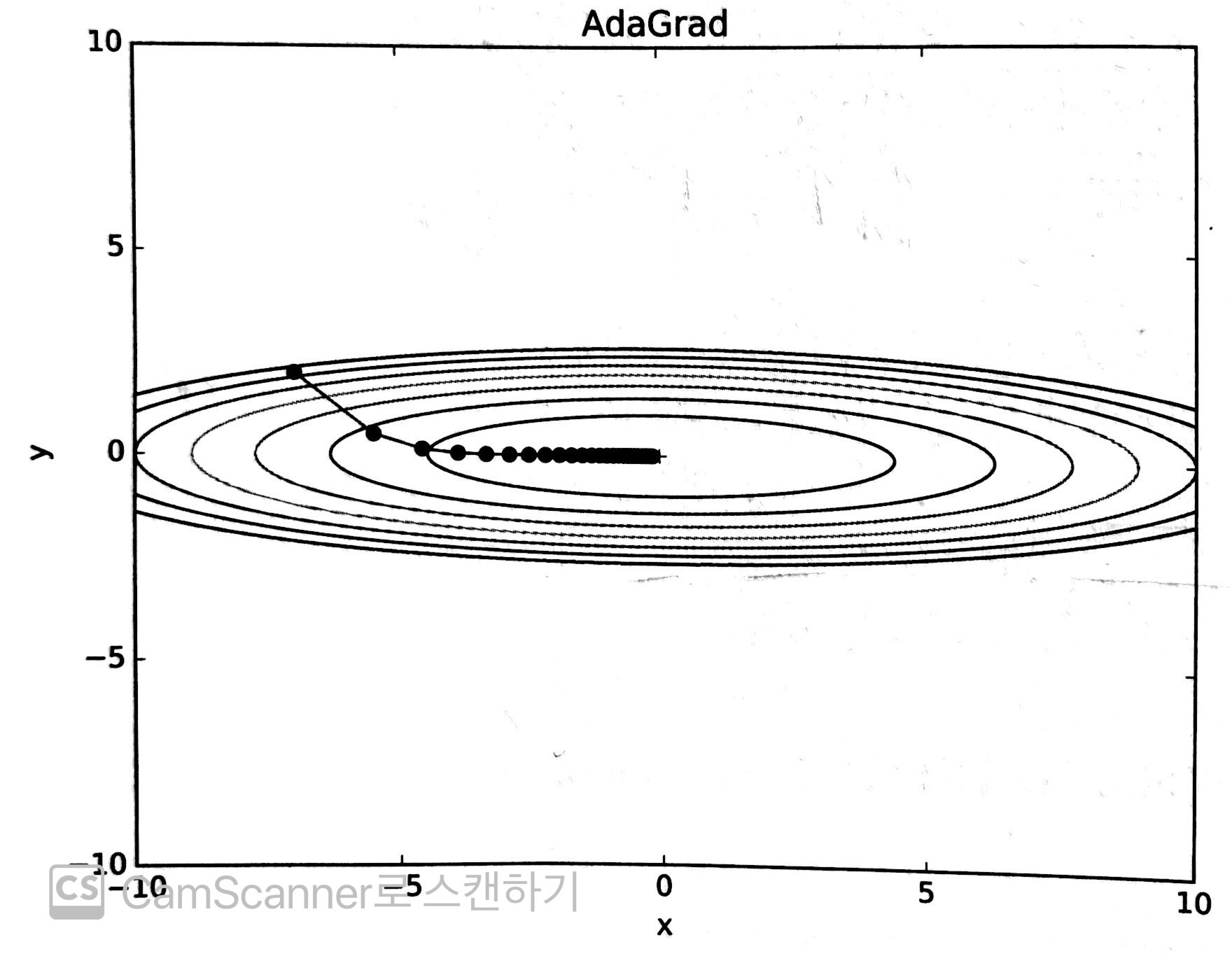
음 확실히 출렁거림이 없어지고, 거의 곧바로 minimum을 찾아감을 볼 수 있다.
그런데 AdaGrad는 계산 과정에서 과거의 기울기가 제곱되며 h에 남아있기 때문에, 무한히 학습하다보면 어느 순간 갱신량(update)이 0에 수렴하는 일이 벌어진다. 이렇게되면 아무리 학습을 하려해도 제자리 걸음이 되는 것인데..
이를 해결하기 위해 등장한 것이 RMSprop이다.
RMSprop(Root Mean Squared propagation) Optimizer (*)

RMSprop의 식을 AdaGrad와 비교한 식이다. 바뀐 부분은 새로운 h(위 수식에선 g_t)를 계산하는 과정인데, 기존의 g_t에 알파로 표기된 감쇠율(decay rate)을 곱해주고, 새로 계산한 그래디언트의 제곱에는 1 - 알파를 곱해준다.
보통 기본 계산해서 감쇠율은 0.9나 0.99등을 사용하는데, 이것이 반복적으로 곱해지면서 과거의 값은 잊혀지고 최신의 값들이 계산에 더 많이 반영되게 된다.
즉 끝없이 그래디언트를 기억하는 것이 아닌, 최신의 값들 위주로 반영함으로써 기울기가 0으로 사라지는 것을 방지한다. 이 같은 방식을 EWMA(지수가중이동평균; Exponentially Weighted Moving Average)라고 한다.
# RMSprop(Root Mean Squared propagation) 클래스 구현
# AdaGrad의 무한히 학습해 기울기가 0이 되는 것을 개선하기 위해 EMA(지수이동평균)를 사용
class RMSprop:
def __init__(self, lr=0.01, decay_rate = 0.99):
self.lr = lr
self.decay_rate = decay_rate # 과거와 최근의 데이터에 감쇠율이라는 가중치를 둔다
self.h = None
def update(self, params, grads):
if self.h is None: # 아직 한 번도 갱신이 되지 않아 h가 없다면
self.h = {}
for key, val in params.items():
self.h[key] = np.zeros_like(val) # 형상에 맞게 0으로 초기화해 생성
for key in params.keys():
# 과거의 데이터에는 감쇠율을, 최신 그래디언트에는 (1-감쇠율)을 곱한다
# 감쇠율(decay_rate)이 작을수록 최신 기울기가 더 많이 반영된다
self.h[key] *= self.decay_rate
self.h[key] += (1 - self.decay_rate) * grads[key] * grads[key]
params[key] -= self.lr * grads[key] / (np.sqrt(self.h[key]) + 1e-7)
.png)
Adam(Adaptive momentum estimation) Optimizer
지금까지 배웠던 모든 내용을 망라하여 새롭게 (2015년에) 등장한 방식이 Adam이다. 이름부터 멋있다. Adam은 Momentum과 RMSprop 및 Bias Correction을 모두 합친 모델로, 수식이 종전의 것에 비해 상당히 복잡하다.
나도 이건 다 이해하긴 어려워서, 참고 영상으로 대체한다. (나중엔 이해할 수 있겠지?)
# ADAM(ADAptive Momentum Estimation) 클래스 구현
# Momentum과 RMSprop(즉 AdaGrad도 일부 반영됨) 및 Bias Correction을 합친 기법
# 여기부턴 어려워서 이해를 포기.. 나중에 다시 보겠지 뭐..
class Adam:
def __init__(self, lr=0.001, beta1=0.9, beta2=0.999):
self.lr = lr
self.beta1 = beta1
self.beta2 = beta2
self.iter = 0
self.m = None
self.v = None
def update(self, params, grads):
if self.m is None:
self.m, self.v = {}, {}
for key, val in params.items():
self.m[key] = np.zeros_like(val)
self.v[key] = np.zeros_like(val)
self.iter += 1
lr_t = self.lr * np.sqrt(1.0 - self.beta2**self.iter) / (1.0 - self.beta1**self.iter)
for key in params.keys():
#self.m[key] = self.beta1*self.m[key] + (1-self.beta1)*grads[key]
#self.v[key] = self.beta2*self.v[key] + (1-self.beta2)*(grads[key]**2)
self.m[key] += (1 - self.beta1) * (grads[key] - self.m[key])
self.v[key] += (1 - self.beta2) * (grads[key]**2 - self.v[key])
params[key] -= lr_t * self.m[key] / (np.sqrt(self.v[key]) + 1e-7)
#unbias_m += (1 - self.beta1) * (grads[key] - self.m[key]) # correct bias
#unbisa_b += (1 - self.beta2) * (grads[key]*grads[key] - self.v[key]) # correct bias
#params[key] += self.lr * unbias_m / (np.sqrt(unbisa_b) + 1e-7)
아담의 핵심은 저 beta1(1차 관성 계수)과 beta2(2차 관성 계수)라고 한다.

AdaGrad처럼 직선형인것보단 Momentum처럼 흔들리는 모양세인데, 그 흔들림의 크기가 작다.
그래서 Optimizer로 뭘 쓰지?
# SGD, Momentum, AdaGrad, Adam의 최적화 기법 비교
# ch06/optimizer_compare_naive.py
# 결과를 보면 AdaGrad가 가장 나아보이긴 하지만, 각 문제와 하이퍼파라미터 설정 등에 따라 달라질 수 있음
import numpy as np
import matplotlib.pyplot as plt
from collections import OrderedDict
def f(x, y):
return x**2 / 20.0 + y**2
def df(x, y):
return x / 10.0, 2.0*y
init_pos = (-7.0, 2.0)
params = {}
params['x'], params['y'] = init_pos[0], init_pos[1]
grads = {}
grads['x'], grads['y'] = 0, 0
optimizers = OrderedDict()
optimizers["SGD"] = SGD(lr=0.95)
optimizers["Momentum"] = Momentum(lr=0.1)
optimizers["AdaGrad"] = AdaGrad(lr=1.5)
optimizers["Adam"] = Adam(lr=0.3)
idx = 1
for key in optimizers:
optimizer = optimizers[key]
x_history = []
y_history = []
params['x'], params['y'] = init_pos[0], init_pos[1]
for i in range(30):
x_history.append(params['x'])
y_history.append(params['y'])
grads['x'], grads['y'] = df(params['x'], params['y'])
optimizer.update(params, grads)
x = np.arange(-10, 10, 0.01)
y = np.arange(-5, 5, 0.01)
X, Y = np.meshgrid(x, y)
Z = f(X, Y)
# 외곽선 단순화
mask = Z > 7
Z[mask] = 0
# 그래프 그리기
plt.subplot(2, 2, idx)
idx += 1
plt.plot(x_history, y_history, 'o-', color="red")
plt.contour(X, Y, Z)
plt.ylim(-10, 10)
plt.xlim(-10, 10)
plt.plot(0, 0, '+')
#colorbar()
#spring()
plt.title(key)
plt.xlabel("x")
plt.ylabel("y")
plt.show()

이것만 놓고보면 AdaGrad가 가장 좋아 보이지만, 문제 상황과 하이퍼파라미터 값등에 따라 달라진다고 한다. 보통 SGD와 Adam이 주로 쓰인다고
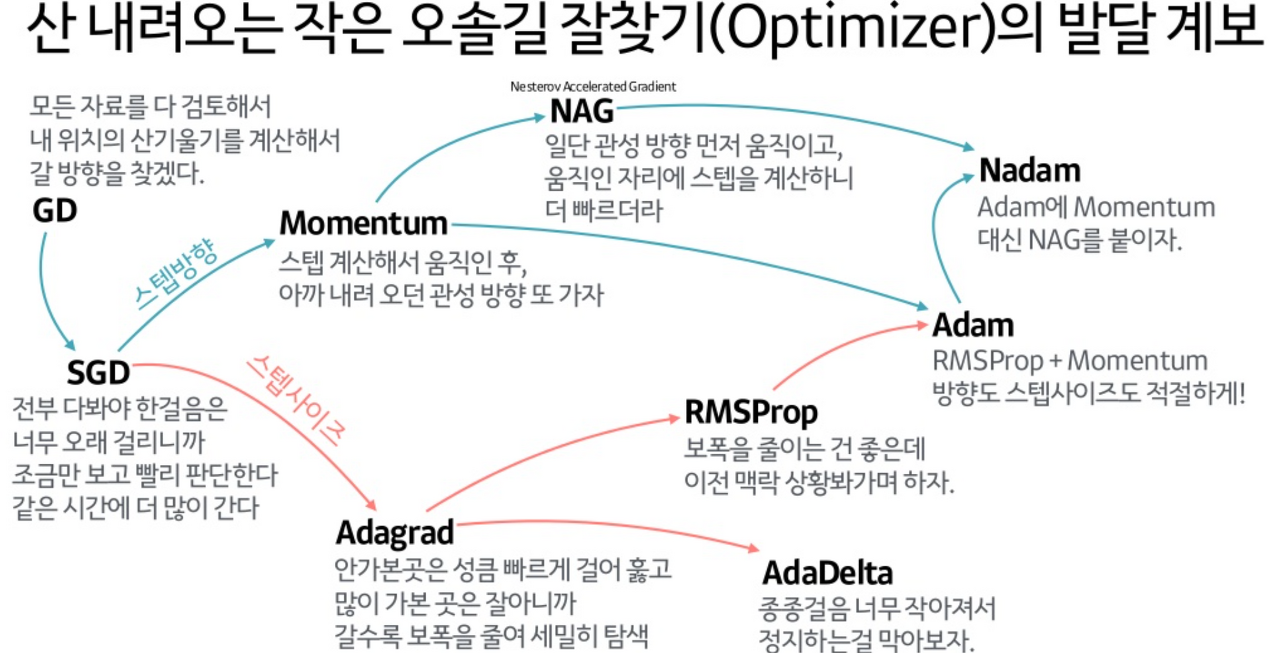
지금까지 살펴본 걸 요약하면 다음과 같다. 추가로 MNIST 데이터셋을 통해 4개 Optimizer의 성능을 비교해보면
# SGD, Momentum, AdaGrad, Adam의 MNIST 데이터셋 비교
# ch06/optimizer_compare_mnist.py
# SGD는 학습 진도가 가장 느려 Loss 값이 늦게 줄어 듦
# 나머지 셋은 고만고만한데, 여기선 AdaGrad가 가장 성능이 좋음
import os, sys
sys.path.append('/content/drive/<여기에 주소 입력>') # 내가 저장한 구글 드라이브 위치
import matplotlib.pyplot as plt
# common 파일과 mnist.py: 옮긴이 깃허브 -> 내 작업 파일로 이동
from common.util import smooth_curve
from common.multi_layer_net import MultiLayerNet
from mnist import load_mnist
# 0. MNIST 데이터 읽기==========
(x_train, t_train), (x_test, t_test) = load_mnist(normalize=True)
train_size = x_train.shape[0]
batch_size = 128
max_iterations = 2000
# 1. 실험용 설정==========
optimizers = {}
optimizers['SGD'] = SGD()
optimizers['Momentum'] = Momentum()
optimizers['AdaGrad'] = AdaGrad()
optimizers['Adam'] = Adam()
#optimizers['RMSprop'] = RMSprop()
networks = {}
train_loss = {}
for key in optimizers.keys():
networks[key] = MultiLayerNet(
input_size=784, hidden_size_list=[100, 100, 100, 100],
output_size=10)
train_loss[key] = []
# 2. 훈련 시작==========
for i in range(max_iterations):
batch_mask = np.random.choice(train_size, batch_size)
x_batch = x_train[batch_mask]
t_batch = t_train[batch_mask]
for key in optimizers.keys():
grads = networks[key].gradient(x_batch, t_batch)
optimizers[key].update(networks[key].params, grads)
loss = networks[key].loss(x_batch, t_batch)
train_loss[key].append(loss)
if i % 100 == 0:
print( "===========" + "iteration:" + str(i) + "===========")
for key in optimizers.keys():
loss = networks[key].loss(x_batch, t_batch)
print(key + ":" + str(loss))
# 3. 그래프 그리기==========
markers = {"SGD": "o", "Momentum": "x", "AdaGrad": "s", "Adam": "D"}
x = np.arange(max_iterations)
for key in optimizers.keys():
plt.plot(x, smooth_curve(train_loss[key]), marker=markers[key], markevery=100, label=key)
plt.xlabel("iterations")
plt.ylabel("loss")
plt.ylim(0, 1)
plt.legend()
plt.show()

역시나 AdaGrad가 가장 좋지만 Momentum과 Adam도 좋은 편이고, SGD만이 상당히 저조한 성적을 보여준다.
가중치 감소(Weight Decay)
가중치를 감소시키자는 아이디어는 오버피팅(overfitting; 과적합)을 방지하기 위해 등장했다. 점차 가중치 w를 감소시켜야 어느정도의 룸(여유 공간? 기존과 다른 테스트 데이터가 들어와도 처리할 수 있는 유연성)을 둘 수 있기 때문
그런데 가중치를 최초에 전부 0으로 세팅하면은 또 안 된다. 정확히는 ‘가중치가 다 같은 값을 띠면 안된다’. 각 노드별로 차이가 생기지 않기에, 노드를 여러개를 둬도 하나로 합치는 것과 다를 바가 없기 때문이다. 이것을 신경망이 대칭성을 띤다(symmetric하다)라고 말한다
그럼 어떻게 해야하는데? 현재 우리가 쓰고 있는 이 코드는 Z(0,1)의 표준정규분포에서 가중치를 랜덤으로 뽑아쓰고 있다. 이걸 1, 0.01, 1/sqrt(n)을 곱해서 결과를 확인해보고 더 이야기를 나눠보자. 활성화함수는 시그모이드다.
Xavier 초깃값(Sigmoid 계열)
# 각 층이 동일한 5층짜리 딥러닝 모델
# 활성화함수는 sigmoid
# 각각 가중치 w의 표준편차가 1, 0.01, 1 / sqrt(n)(Xavier 초깃값)일 때
# * 1: 활성화함수를 통과한 값들이 0과 1쪽에만 분포한다. 기울기 소실(gradient vanishing) 발생!!
# * 0.01: 기울기 소실은 없지만, 활성화값들이 치우쳐져있음. 표현력이 제한되어 뉴런이 하나이니만 못함
# / np.sqrt(n): Xavier 초깃값을 사용하니 활성화값이 적당히 고루 분포하게 바뀌었다!
import numpy as np
import matplotlib.pyplot as plt
def sigmoid(x):
return 1 / (1 + np.exp(-x))
for j in range(3):
input_data = np.random.randn(1000, 100) # 1000개의 데이터
node_num = 100 # 각 은닉층의 노드(뉴런) 수
hidden_layer_size = 5 # 은닉층이 5개
activations = {} # 이곳에 활성화 결과를 저장
x = input_data
for i in range(hidden_layer_size):
if i != 0:
x = activations[i-1]
# 초깃값을 다양하게 바꿔가며 실험해보자!
if (j == 0):
w = np.random.randn(node_num, node_num) * 1
elif (j == 1):
w = np.random.randn(node_num, node_num) * 0.01
else:
w = np.random.randn(node_num, node_num) / np.sqrt(node_num)
a = np.dot(x, w)
# 활성화 함수도 바꿔가며 실험해보자!
z = sigmoid(a)
activations[i] = z
# 히스토그램 그리기
for i, a in activations.items():
plt.subplot(1, len(activations), i+1)
plt.title(str(i+1) + "-layer")
if i != 0: plt.yticks([], [])
plt.hist(a.flatten(), 30, range=(0,1))
plt.show()
먼저 그대로 랜덤값을 사용할 때, 즉 1을 곱했을 때다.

층을 통과할수록 활성화값들이 0과 1쪽으로 모이게 되는데, 이를 기울기 소실(gradient vanishing)이라고 한다. 딥러닝 쪽에서 아주 명성높은 문제점이다.
이것은 우리가 시그모이드 계열 함수를 사용했기 때문에 발생하는데, 시그모이드 계열은 양 끝으로 갈수록 함수의 기울기가 줄어들어 미분값이 0에 수렴하게 된다. 따라서 역전파의 기울기 값도 줄고, 학습 과정에서의 업데이트도 사라지게 된다. 성능은 당연히 안 좋아진다.

반대로 0.01을 곱해보니 이번엔 활성화값이 가운데로 모여버렸다. 이것도 안 좋은 모양세이다. 활성화값들이 치우쳐질 수록 표현력이 제한돼 뉴런을 많이 쓴 들, 층의 개수를 늘린 들 무용지물이 되어버린다.
각 층의 활성화값은 적당히 고루 분포해야한다. 층 사이에 다양한 방향에서 데이터가 흘러야 망이 활성화되는데, 각 노드별로 특성이 없이 다 비슷하다면 망이 있는들 큰 의미가 없어지기 때문이다.

어 그런데 1 / sqrt(n)을 곱하니 적당히 종형 분포가 나오면서 활성화값들이 충분히 산개되었다! 이것은 1 / sqrt(n) 값이 Xavier 초깃값이기 때문이다.
이 Xavier(사비에르) 초깃값은 시그모이드 계열처럼 활성화 함수가 (특히, 원점에서) 선형을 띨 때 그 진가를 발휘하는 초깃값이다. 최초 가중치의 표준편차를 1 / sqrt(n)으로 만드는 것인데, 확실히 활성화값들이 넓게 분포됨을 확인할 수 있다.
# 각 층이 동일한 5층짜리 딥러닝 모델
# 활성화함수는 tanh, 가중치 w의 표준편차가 1 / sqrt(n)일 때
# sigmoid에 비해 tanh는 기함수라 활성화값이 좀 더 말끔하다는데... 말끔한가..?
import numpy as np
import matplotlib.pyplot as plt
def tanh(x):
return np.tanh(x)
input_data = np.random.randn(1000, 100) # 1000개의 데이터
node_num = 100 # 각 은닉층의 노드(뉴런) 수
hidden_layer_size = 5 # 은닉층이 5개
activations = {} # 이곳에 활성화 결과를 저장
x = input_data
for i in range(hidden_layer_size):
if i != 0:
x = activations[i-1]
# 초깃값을 다양하게 바꿔가며 실험해보자!
w = np.random.randn(node_num, node_num) / np.sqrt(node_num)
a = np.dot(x, w)
# 활성화 함수도 바꿔가며 실험해보자!
z = tanh(a)
activations[i] = z
# 히스토그램 그리기
for i, a in activations.items():
plt.subplot(1, len(activations), i+1)
plt.title(str(i+1) + "-layer")
if i != 0: plt.yticks([], [])
# plt.xlim(0.1, 1)
# plt.ylim(0, 7000)
plt.hist(a.flatten(), 30, range=(0,1))
plt.show()
번외로, 시그모이드 계열의 또 다른 대표인 tanh(탄젠트 하이퍼볼릭)을 사용하면 sigmoid보다 더 좋다더라..? 이는 tanh가 원점대칭인 기함수이기 때문인데, 실제 그래프를 보니..? 어 그런가? 방금 전 종형 그래프에 비해서는 잘 모르겠는데.. 아무튼 그렇다더라

Xavier 초깃값 유도 (*)
어느 통계학 문제가 그렇듯, 가정이 필요하다.
먼저 활성 함수인 sigmoid계열이 선형성(linearity)을 띤다고 가정하자(원점 근처에선 선형성을 띠니 얼추 맞다). 또 입력 데이터 x와 가중치 w는 서로 같은 차원의 분포이며 독립인 iid(independent and identically distributed)라고 가정하자. 이때 x와 w는 표준정규분포에서 나왔으므로 각각의 평균(기댓값)은 0이다.
먼저 활성화 함수 y는 x와 w의 선형변환(linear transformation)이라고 가정했으므로 다음과 같이 작성된다.
\[y=w_1 x_1+w_2 x_2+...+w_n x_n+b\]이후 분산을 계산하는데, x와 w는 iid이므로 각 식을 나눌 수 있다.
\[\begin{align} Var(y)&=Var(w_1 x_1+w_2 x_2+...+w_n x_n+b) \\ &=Var(w_1 x_1)+Var(w_2 x_2)+...+Var(w_n x_n) \\ &=\sum_{i=1}^{n}Var(w_i x_i)\end{align}\]이때
\[E(w_i)=0, E(x_i)=0 \Rightarrow Var(x_i w_i)=Var(x_i)Var(w_i)\]이므로
\[\begin{align} Var(y)&=\sum_{i=1}^{n}E((w_i x_i)^2)-E(w_i x_i)^2 \\ &=\sum_{i=1}^{n}E(w_i^2)E(x_i^2)-E(w_i)^2E(x_i)^2 \end{align}\]여기서 분산은 ‘(제곱의 평균) - (평균의 제곱)’이라는 식을 변형하면
\[\] \[\begin{align} &=\sum_{i=1}^{n}(Var(w_i)+E(w_i)^2)(Var(x_i)+E(x_i)^2)-E(w_i)^2E(x_i)^2 \\ &=\sum_{i=1}^{n}E(w_i)^2Var(x_i)+E(x_i)^2Var(w_i)+Var(x_i)Var(w_i) \\ &=\sum_{i=1}^{n}Var(x_i)Var(w_i) \end{align}\]이때 x_i와 w_i의 분포가 모두 같고, 가중치 초기화를 통해 입력과 출력의 분산을 같게 만들려면 Var(x_i) = Var(y)이므로
\[=nVar(x_i)Var(w_i)=nVar(y)Var(w_i)\]에서 양변을 n으로 나누어 정리하면
\[Var(w_i)=\frac{1}{n}\]즉 가중치의 분산은 1 / n 이므로, 가중치의 표준편차인 Xavier 초깃값은 1 / sqrt(n)이다.
\[\sigma (w_i)=\frac{1}{\sqrt{n}}\]He 초깃값(ReLU 계열)
Xavier 초깃값이 사비에르 교수의 이름을 땄다면, He 초깃값은 히 교수의 이름을 땄다. 이 초깃값은 앞선 sigmoid 계열이 아닌 ReLU 계열의 활성화함수를 사용할 때 유용하다고 한다.
Xavier 초깃값이 1 / sqrt(n)이었다면, He 초깃값은 sqrt(2 / n)이다. 코드부터 확인해보자.
# 각 층이 동일한 5층짜리 딥러닝 모델
# 활성화함수는 ReLU
# 각각 가중치 w의 표준편차가 0.01, 1 / sqrt(n)(Xavier 초깃값), sqrt(2 / n)(He 초깃값)일 때
# ReLU에 특화된 He 초깃값에서 활성화값의 분포가 가장 균일하다
import numpy as np
import matplotlib.pyplot as plt
def ReLU(x):
return np.maximum(0, x)
for j in range(3):
input_data = np.random.randn(1000, 100) # 1000개의 데이터
node_num = 100 # 각 은닉층의 노드(뉴런) 수
hidden_layer_size = 5 # 은닉층이 5개
activations = {} # 이곳에 활성화 결과를 저장
x = input_data
for i in range(hidden_layer_size):
if i != 0:
x = activations[i-1]
# 초깃값을 다양하게 바꿔가며 실험해보자!
if (j == 0):
w = np.random.randn(node_num, node_num) * 0.01
elif (j == 1):
w = np.random.randn(node_num, node_num) * np.sqrt(1.0 / node_num)
else:
w = np.random.randn(node_num, node_num) * np.sqrt(2.0 / node_num)
a = np.dot(x, w)
# 활성화 함수도 바꿔가며 실험해보자!
z = ReLU(a)
activations[i] = z
# 히스토그램 그리기
for i, a in activations.items():
plt.subplot(1, len(activations), i+1)
plt.title(str(i+1) + "-layer")
if i != 0: plt.yticks([], [])
plt.xlim(0, 1)
plt.ylim(0, 7000)
plt.hist(a.flatten(), 30, range=(0,1))
plt.show()

먼저 ReLU 함수에 표준편차를 0.01로 잡았을 때다. 택도 없다.

오 역시 Xavier 초깃값은 괜찮은 편이다. 그러나 층을 거듭할 수록 점점 0쪽으로 활성화값들이 치우쳐지며 기울기 소실의 우려가 있어 보인다.

그러나 우리의 He 초깃값은 실망시키지 않는다. 정말 균등하게 잘 분포되어있는 것을 볼 수 있다. 따라서 결론은 활성화 함수로 RELU 계열을 사용할 때는 He 초깃값을, sigmoid 계열을 사용할 때는 Xavier 초깃값을 사용하면 된다는 것이다.
He 초깃값 유도 (*)
참고한 책에는, ReLU를 사용하면 출력의 분산이 절반으로 줄기 때문에, 분산을 2배로 늘려서 모델링을 맞춘다고 설명이 되어있다. 결론은
\[Var(w)=\frac{2}{n}, \sigma (w_i)=\sqrt{\frac{2}{n}}\]그래서 초깃값으로 뭘 쓰지?
말했잖아 위에서. 아니 글을 안 읽니? (후우)
장난이고 코드부터 확인해보자.
# 각 층이 동일한 5층짜리 딥러닝 모델로 MNIST 데이터셋을 학습 시킴
# 활성화함수는 ReLU로만 고정
# 각각 가중치 w의 표준편차가 0.01, 1 / sqrt(n)(Xavier 초깃값), 2 / sqrt(n)(He 초깃값)일 때를 비교
# 역시나 ReLU에 특화된 He 초깃값에서 loss가 가장 적다.
# ch06/weight_init_compare.py
import os, sys
import numpy as np
sys.path.append('/content/drive/<여기에 주소 입력>') # 내가 저장한 구글 드라이브 위치
import matplotlib.pyplot as plt
# common 파일과 mnist.py: 옮긴이 깃허브 -> 내 작업 파일로 이동
from mnist import load_mnist
from common.util import smooth_curve
from common.multi_layer_net import MultiLayerNet
from common.optimizer import SGD
# 0. MNIST 데이터 읽기==========
(x_train, t_train), (x_test, t_test) = load_mnist(normalize=True)
train_size = x_train.shape[0]
batch_size = 128
max_iterations = 2000
# 1. 실험용 설정==========
weight_init_types = {'std=0.01': 0.01, 'Xavier': 'sigmoid', 'He': 'relu'}
optimizer = SGD(lr=0.01)
networks = {}
train_loss = {}
for key, weight_type in weight_init_types.items():
networks[key] = MultiLayerNet(input_size=784, hidden_size_list=[100, 100, 100, 100],
output_size=10, weight_init_std=weight_type)
# 파라미터 weight_init_std가 MultiLayerNet.__init_weight로 넘어가서 가중치 표준편차를 정함
train_loss[key] = []
# 2. 훈련 시작==========
for i in range(max_iterations):
batch_mask = np.random.choice(train_size, batch_size)
x_batch = x_train[batch_mask]
t_batch = t_train[batch_mask]
for key in weight_init_types.keys():
grads = networks[key].gradient(x_batch, t_batch)
optimizer.update(networks[key].params, grads)
loss = networks[key].loss(x_batch, t_batch)
train_loss[key].append(loss)
if i % 100 == 0:
print("===========" + "iteration:" + str(i) + "===========")
for key in weight_init_types.keys():
loss = networks[key].loss(x_batch, t_batch)
print(key + ":" + str(loss))
# 3. 그래프 그리기==========
markers = {'std=0.01': 'o', 'Xavier': 's', 'He': 'D'}
x = np.arange(max_iterations)
for key in weight_init_types.keys():
plt.plot(x, smooth_curve(train_loss[key]), marker=markers[key], markevery=100, label=key)
plt.xlabel("iterations")
plt.ylabel("loss")
plt.ylim(0, 2.5)
plt.legend()
plt.show()

ReLU를 사용해서 그런지 He가 잘 먹힌다. 물론 Xavier도 선방하는 편. 표준편차가 0.01인 것은 답도 없다. 위에서 알려준대로 활성화함수 계열에 맞춰서 잘 사용하자.
배치 정규화(Batch Normalization)
방금 전까지는 초깃값을 만져서 최대한 활성화값 분포가 퍼지도록 유도했지만, 우리가 ‘강제로’ 분산되게끔 유도할 수도 있다. 종형 분포를 만들 때 매우 쉬운 방법 중 하나인, 정규화를 사용하는 것이다.
\[\mu _B\leftarrow \frac{1}{m} \sum_{i=1}^{m}x_i\] \[\sigma_B^2\leftarrow \frac{1}{m} \sum_{i=1}^{m}(x_i-\mu_B)^2\] \[\widehat{x}_i\leftarrow \frac{x_i-\mu_B}{\sqrt{\sigma_B^2}+\varepsilon }\]고등학교 확률과통계 시간에 배운 방법 그대로 실시하면 된다. 분산은 편차 제곱의 평균으로 구한 후, 엡실론(아주 작은 값, 보통 1-e7)과 더해 ‘변수 빼기 평균 나누기 표준편차’로 정규화한다.
\[y_i\leftarrow \gamma \widehat{x}_i+\beta\]여기에 감마(gamma)로 확대에 대한 조정을, 베타(beta)로 이동에 대한 조정을 실시하면 배치 정규화가 된다.
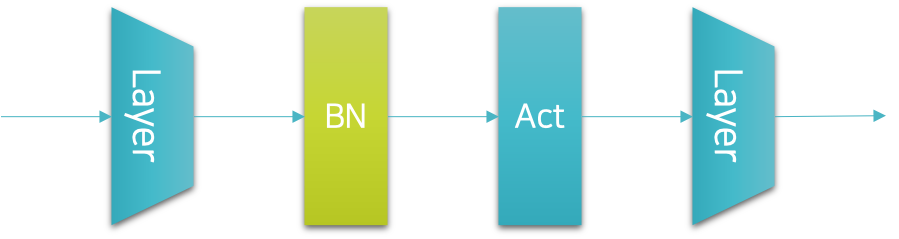
배치 정규화 계층은 세르게이 이오페 박사에게 처음 제안될 당시, 활성화 계층 전에 삽입되는 것으로 구현됐었다. 그러나 요즘은 활성화 함수 뒤에 넣는 추세라고..! 역전파 유도는 복잡하다고 하니 내 수준에선 넘어가겠다..
# bn: batch normalization: 활성화 함수 계층 전후에 정규화(normalization: Z(0, 1)) 계층을 집어 넣은 것
# 대체적으로 배치 정규화를 사용한 실선(파란색)이 그렇지 않은 점선(주황색)보다 정확도가 높다!
# ch06/batch_norm_test.py
import sys, os
sys.path.append('/content/drive/<여기에 주소 입력>') # 내가 저장한 구글 드라이브 위치
import numpy as np
import matplotlib.pyplot as plt
# common 파일과 mnist.py: 옮긴이 깃허브 -> 내 작업 파일로 이동
from mnist import load_mnist
from common.multi_layer_net_extend import MultiLayerNetExtend
from common.optimizer import SGD, Adam
(x_train, t_train), (x_test, t_test) = load_mnist(normalize=True)
# 학습 데이터를 줄임
x_train = x_train[:1000]
t_train = t_train[:1000]
max_epochs = 20
train_size = x_train.shape[0]
batch_size = 100
learning_rate = 0.01
# 그래프 그리기 과정에서 실행하는 __train 함수
def __train(weight_init_std):
bn_network = MultiLayerNetExtend(input_size=784, hidden_size_list=[100, 100, 100, 100, 100], output_size=10,
weight_init_std=weight_init_std, use_batchnorm=True) # bn = True!
network = MultiLayerNetExtend(input_size=784, hidden_size_list=[100, 100, 100, 100, 100], output_size=10,
weight_init_std=weight_init_std)
optimizer = SGD(lr=learning_rate)
train_acc_list = []
bn_train_acc_list = []
iter_per_epoch = max(train_size / batch_size, 1)
epoch_cnt = 0
for i in range(1000000000): # 데이터를 줄인 대신 반복을 10억 회로 늘림
batch_mask = np.random.choice(train_size, batch_size)
x_batch = x_train[batch_mask]
t_batch = t_train[batch_mask]
for _network in (bn_network, network):
grads = _network.gradient(x_batch, t_batch)
optimizer.update(_network.params, grads)
if i % iter_per_epoch == 0:
train_acc = network.accuracy(x_train, t_train)
bn_train_acc = bn_network.accuracy(x_train, t_train)
train_acc_list.append(train_acc)
bn_train_acc_list.append(bn_train_acc)
print("epoch:" + str(epoch_cnt) + " | " + str(train_acc) + " - " + str(bn_train_acc))
epoch_cnt += 1
if epoch_cnt >= max_epochs:
break
return train_acc_list, bn_train_acc_list
# 그래프 그리기==========
weight_scale_list = np.logspace(0, -4, num=16) # w 값으로 1에서 0.0001까지. 진수 [0, -4]를 16등분한 계산
x = np.arange(max_epochs)
for i, w in enumerate(weight_scale_list):
print( "============== " + str(i+1) + "/16" + " ==============")
train_acc_list, bn_train_acc_list = __train(w)
plt.subplot(4,4,i+1)
plt.title("W:" + str(w))
if i == 15:
plt.plot(x, bn_train_acc_list, label='Batch Normalization', markevery=2)
plt.plot(x, train_acc_list, linestyle = "--", label='Normal(without BatchNorm)', markevery=2)
else:
plt.plot(x, bn_train_acc_list, markevery=2)
plt.plot(x, train_acc_list, linestyle="--", markevery=2)
plt.ylim(0, 1.0)
if i % 4:
plt.yticks([])
else:
plt.ylabel("accuracy")
if i < 12:
plt.xticks([])
else:
plt.xlabel("epochs")
plt.legend(loc='lower right')
plt.show()

전반적으로 배치 정규화를 한 파란색 실선이, 그렇지 않은 주황색 점선보다 정확도가 높은 것을 볼 수 있다.
오버피팅(Overfitting) 방지
이번엔 과적합(오버피팅)을 방지하는 방법에 대해 논해보겠다. 과적합은 매개변수가 너무 많거나 훈련 데이터가 적어, 해당 데이터에만 몰두하고 유연성이 없을 때 발생한다. 먼저 의도적으로 오버피팅을 재현해보겠다.
# 의도적으로 오버피팅을 재현
# 훈련 데이터를 줄이고 복잡한 7층 네트워크를 사용
# 135회 에포크만에 훈련 데이터는 100% 정확도를 보이지만
# 200회 에포크가 다 끝나도 테스트 데이터는 75%의 정확도만을 보임 -> 오버피팅
# ch06/overfit_weight_decay.py
import os, sys
sys.path.append('/content/drive/<여기에 주소 입력>') # 내가 저장한 구글 드라이브 위치
import numpy as np
import matplotlib.pyplot as plt
# common 파일과 mnist.py: 옮긴이 깃허브 -> 내 작업 파일로 이동
from mnist import load_mnist
from common.multi_layer_net import MultiLayerNet
from common.optimizer import SGD
(x_train, t_train), (x_test, t_test) = load_mnist(normalize=True)
# 오버피팅을 재현하기 위해 학습 데이터 수를 줄임
x_train = x_train[:300]
t_train = t_train[:300]
# weight decay(가중치 감쇠) 설정 =======================
weight_decay_lambda = 0 # weight decay를 사용하지 않을 경우
#weight_decay_lambda = 0.1
# ====================================================
# 오버피팅을 재현하기 위해 hidden_size_list를 늘림
network = MultiLayerNet(input_size=784, hidden_size_list=[100, 100, 100, 100, 100, 100], output_size=10,
weight_decay_lambda=weight_decay_lambda)
optimizer = SGD(lr=0.01) # 학습률이 0.01인 SGD로 매개변수 갱신
max_epochs = 201
train_size = x_train.shape[0]
batch_size = 100
train_loss_list = []
train_acc_list = []
test_acc_list = []
iter_per_epoch = max(train_size / batch_size, 1)
epoch_cnt = 0
for i in range(1000000000):
batch_mask = np.random.choice(train_size, batch_size)
x_batch = x_train[batch_mask]
t_batch = t_train[batch_mask]
grads = network.gradient(x_batch, t_batch)
optimizer.update(network.params, grads)
if i % iter_per_epoch == 0:
train_acc = network.accuracy(x_train, t_train)
test_acc = network.accuracy(x_test, t_test)
train_acc_list.append(train_acc)
test_acc_list.append(test_acc)
print("epoch:" + str(epoch_cnt) + ", train acc:" + str(train_acc) + ", test acc:" + str(test_acc))
epoch_cnt += 1
if epoch_cnt >= max_epochs:
break
# 그래프 그리기==========
markers = {'train': 'o', 'test': 's'}
x = np.arange(max_epochs)
plt.plot(x, train_acc_list, marker='o', label='train', markevery=10)
plt.plot(x, test_acc_list, marker='s', label='test', markevery=10)
plt.xlabel("epochs")
plt.ylabel("accuracy")
plt.ylim(0, 1.0)
plt.legend(loc='lower right')
plt.show()

문제점이 2가지가 보이는데, 첫째는 훈련 데이터와 테스트 데이터에서 25% 정도의 정확도 차이가 발생한다는 것이고, 둘째는 훈련 데이터셋에서 100%의 정확도가 나와버렸다는 것이다. 둘다 범용성이 없음을 방증한다. 즉 훈련 데이터에만 적응(fitting)해버린 과적합 상황인 것
가중치 감소
그렇다면 앞서 Xavier/He를 논할 때 말했던 가중치 감소(weight decay)를 적용해보겠다. 아래의 결과는 감쇠율을 10%로 바꾸고(위 코드의 # weight decay 파트 참조) 다시 정확도를 계산한 것이다.

최종 훈련 데이터 정확도가 87%, 테스트 데이터가 70%이다. 오버피팅도 방지했고, 훈련 데이터셋이 100%가 아니라 범용성도 있음을 알 수 있다.
드롭아웃(Dropout)
이외에도 뉴런을 임의로 삭제하면서 학습하는 방식인 드롭아웃(dropout) 방식이 존재한다. 마치 우리 뇌의 뉴런에서 자주 사용하지 않는 시냅스 쪽은 퇴화하는 방식을 차용한 듯 한데, 이건 노드를 ‘무작위로’ 선택해 삭제한다는 차이가 있다.
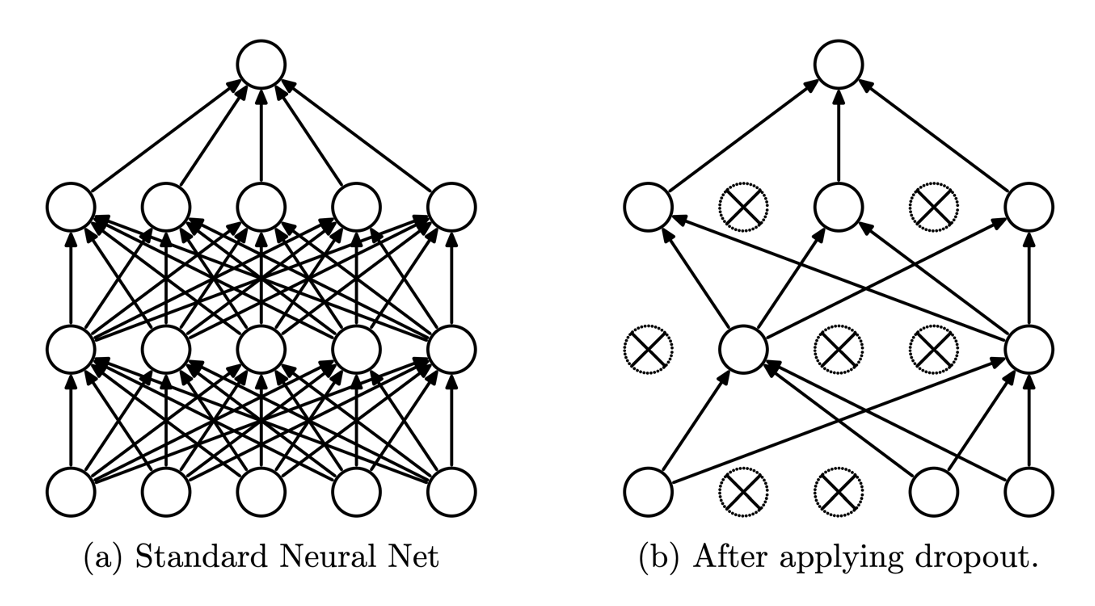
# 드롭아웃(Dropout) 구현
class Dropout:
def __init__(self, dropout_ratio=0.5): # 보통 50%로 설정. 타노스야?
self.dropout_ratio = dropout_ratio
self.mask = None
def forward(self, x, train_flg=True):
if train_flg:
self.mask = np.random.rand(*x.shape) > self.dropout_ratio
return x * self.mask
else:
return x * (1.0 - self.dropout_ratio)
def backward(self, dout):
return dout * self.mask
책에 나온 내용으로는 랜덤으로 생성된 값이 dropout_ratio보다 큰 것만 True로 masking을 한다는데, 다른 블로그에서는 삭제되는 뉴런의 비율이라는 말도 있고.. 코드마다 조금씩 방식이 다른 걸까?
# 드롭아웃을 MNIST 데이터셋에 적용
# 상당히 정확도가 낮게 나오지만, 일단 오버피팅 방지와 범용성 확보가 됨
# 드롭아웃율을 잘 조절해야할 듯
# ch06/overfit_dropout.py
import os, sys
sys.path.append('/content/drive/<여기에 주소 입력>') # 내가 저장한 구글 드라이브 위치
import numpy as np
import matplotlib.pyplot as plt
# common 파일과 mnist.py: 옮긴이 깃허브 -> 내 작업 파일로 이동
from mnist import load_mnist
from common.multi_layer_net_extend import MultiLayerNetExtend
from common.trainer import Trainer
(x_train, t_train), (x_test, t_test) = load_mnist(normalize=True)
# 오버피팅을 재현하기 위해 학습 데이터 수를 줄임
x_train = x_train[:300]
t_train = t_train[:300]
# 드롭아웃 사용 유무와 비울 설정 ========================
use_dropout = True # 드롭아웃을 쓰지 않을 때는 False
dropout_ratio = 0.2
# ====================================================
network = MultiLayerNetExtend(input_size=784, hidden_size_list=[100, 100, 100, 100, 100, 100],
output_size=10, use_dropout=use_dropout, dropout_ration=dropout_ratio)
trainer = Trainer(network, x_train, t_train, x_test, t_test,
epochs=301, mini_batch_size=100,
optimizer='sgd', optimizer_param={'lr': 0.01}, verbose=True)
trainer.train()
train_acc_list, test_acc_list = trainer.train_acc_list, trainer.test_acc_list
# 그래프 그리기==========
markers = {'train': 'o', 'test': 's'}
x = np.arange(len(train_acc_list))
plt.plot(x, train_acc_list, marker='o', label='train', markevery=10)
plt.plot(x, test_acc_list, marker='s', label='test', markevery=10)
plt.xlabel("epochs")
plt.ylabel("accuracy")
plt.ylim(0, 1.0)
plt.legend(loc='lower right')
plt.show()

테스트 데이터에선 최종 49.2%의 정확도가 나오긴 했지만, 이건 dropout_ratio를 조정하면 될 일인 듯하다.
하이퍼파라미터 최적화
이제 적당한 범위 내에서 하이퍼파라미터를 설정해서 뭐가 좋은지 직접 다 돌려볼건데, 여기서 검증 데이터(validation data)라는 개념이 나온다.
쉽게 말해 학습 데이터 중 일부(여기선 20%)를 떼어내서 하이퍼파라미터를 검증 데이터로 평가하고, 이 전체 모델을 테스트 데이터로 (최종적으로 한 번만) 성능 평가를 실시하겠다는 내용이다.
하이퍼파라미터의 범위는 로그 스케일(log scale: 10의 거듭제곱 단위)로 설정하면 좋다고 알려져있다. 원래는 이게 정말 시간이 오래 걸리는 작업이지만, 우리가 만든 간단한 모델에서 먼저 적용해보자.
# 가중치 감소 계수(weight_decay)를 10^-8~10^-4, 학습률(lr)을 10^-6~10^-2로 설정
# 최고 정확도 84%, 교재대로 lr은 0.001~0.01에서, 가중치 감소 계수는 10의 -8승~-6승에서 학습이 잘 되는 듯
import os, sys
sys.path.append('/content/drive/<여기에 주소 입력>') # 내가 저장한 구글 드라이브 위치
import numpy as np
import matplotlib.pyplot as plt
# common 파일과 mnist.py: 옮긴이 깃허브 -> 내 작업 파일로 이동
from mnist import load_mnist
from common.multi_layer_net import MultiLayerNet
from common.util import shuffle_dataset
from common.trainer import Trainer
(x_train, t_train), (x_test, t_test) = load_mnist(normalize=True)
# 결과를 빠르게 얻기 위해 훈련 데이터를 줄임
x_train = x_train[:500]
t_train = t_train[:500]
# 20%를 검증 데이터로 분할
validation_rate = 0.20
validation_num = int(x_train.shape[0] * validation_rate)
x_train, t_train = shuffle_dataset(x_train, t_train)
x_val = x_train[:validation_num]
t_val = t_train[:validation_num]
x_train = x_train[validation_num:]
t_train = t_train[validation_num:]
def __train(lr, weight_decay, epocs=50):
network = MultiLayerNet(input_size=784, hidden_size_list=[100, 100, 100, 100, 100, 100],
output_size=10, weight_decay_lambda=weight_decay)
trainer = Trainer(network, x_train, t_train, x_val, t_val,
epochs=epocs, mini_batch_size=100,
optimizer='sgd', optimizer_param={'lr': lr}, verbose=False)
trainer.train()
return trainer.test_acc_list, trainer.train_acc_list
# 하이퍼파라미터 무작위 탐색======================================
optimization_trial = 100
results_val = {}
results_train = {}
for _ in range(optimization_trial):
# 탐색한 하이퍼파라미터의 범위 지정===============
weight_decay = 10 ** np.random.uniform(-8, -4)
lr = 10 ** np.random.uniform(-6, -2)
# ================================================
val_acc_list, train_acc_list = __train(lr, weight_decay)
print("val acc:" + str(val_acc_list[-1]) + " | lr:" + str(lr) + ", weight decay:" + str(weight_decay))
key = "lr:" + str(lr) + ", weight decay:" + str(weight_decay)
results_val[key] = val_acc_list
results_train[key] = train_acc_list
# 그래프 그리기========================================================
print("=========== Hyper-Parameter Optimization Result ===========")
graph_draw_num = 20
col_num = 5
row_num = int(np.ceil(graph_draw_num / col_num))
i = 0
for key, val_acc_list in sorted(results_val.items(), key=lambda x:x[1][-1], reverse=True):
print("Best-" + str(i+1) + "(val acc:" + str(val_acc_list[-1]) + ") | " + key)
plt.subplot(row_num, col_num, i+1)
plt.title("Best-" + str(i+1))
plt.ylim(0.0, 1.0)
if i % 5: plt.yticks([])
plt.xticks([])
x = np.arange(len(val_acc_list))
plt.plot(x, val_acc_list)
plt.plot(x, results_train[key], "--")
i += 1
if i >= graph_draw_num:
break
plt.show()

총 100번의 시도 중 정확도가 가장 높은 20개의 그래프를 나타낸 것인데, Best-5까지는 그래프가 좋아 보인다. 최고 정확도는 84%(Best-1). Best-5까지의 하이퍼파라미터를 살피면 다음과 같다.
Best-1(val acc:0.84) | lr:0.00959798755927851, weight decay:6.007876819941433e-08
Best-2(val acc:0.84) | lr:0.009432847967045455, weight decay:3.14378065039467e-07
Best-3(val acc:0.82) | lr:0.0070103709524546275, weight decay:8.475775019369787e-05
Best-4(val acc:0.81) | lr:0.006838455824975194, weight decay:6.852446028568848e-05
Best-5(val acc:0.78) | lr:0.008103165227243787, weight decay:5.0323160035718685e-05
교재대로 lr은 0.001~0.01에서, 가중치 감소 계수는 10의 -8승~-6승에서 학습이 잘 되었다.
출처: [밑바닥부터 시작하는 딥러닝 Chapter 6]
참고: [Do it! 딥러닝 교과서 p.145~171]